| |
Aberration chromosomique
: Toute anomalie dans la structure ou le nombre de chromosomes d'une espèce.
Acentrique
: Se dit d'un chromosome, ou d'une chromatide, dépourvu de centromère.
Acrocentrique
: Se dit d'un chromosome dont le centromère, situé très près de l'extrémité, sépare le chromosome en deux bras : un long et l'autre extrêmement court.
Allèle
ou allélomorphe : Une ou plusieurs versions alternatives d'un même gène, situées au niveau d'un même locus.
Amplification génique
: Multiplication sélective d'un même gène, sans amplification d'autres gènes.
Anaphase
: Une phase de la mitose ou de la méiose I ou II, caractérisée par la migration des deux chromosomes homologues chacun vers un des pôles du fuseau de division cellulaire.
Aneuploïdie
: Anomalie du nombre de chromosomes, caractérisée par la présence d'un ou plusieurs chromosomes en plus ou en moins du jeu chromosomique diploïde normal (2n).
Aneusomie
: Anomalie de structure d'un chromosome, avec perte ou gain de matériel génétique. L'aneusomie par recombinaison désigne la présence d'une duplication ou d'une délétion résultant d'un crossing-over dans une boucle d'inversion.
Autoradiographie :
Mise en évidence photographique de la présence ou de l'absence d'une substance radioactive incorporée lors du métabolisme dans un tissu ou un organe. L'application de la préparation sur le film ou l'émulsion photographique permet de détecter la répartition de la substance radiomarquée.
Autosome
: Tout chromosome à l'exception des chromosomes sexuels X et Y. Les gènes et les segments chromosomiques situés sur les autosomes sont dits autosomiques.
Auxotrophe
: Cellule ou souche cellulaire, dont la croissance dépend de la présence d'une substance donnée dans le milieu nutritif.

Johann Gregor Mendel (1822-1884)

|
Banque de gène
: Une collection de clones cellulaires, dont chaque contient un DNA recombiné différent et dont l'ensemble représente le génome.
Bivalent
: Désigne deux chromosomes homologues appariés pendant la première division méiotique. Le nombre de bivalents 'n' correspond donc au nombre de paires de chromosomes appariés et est égal à la moitié du nombre de chromosomes présents dans les cellules somatiques : diploïdes (2n). Les bivalents constituent les structures cytogénétiques nécessaires aux crossing-over. Lors de la méiose d'une cellule trisomique, les trois chromosomes forment un trivalent.
|
CAAT-Box
: Séquence de DNA régulatrice, située dans la région 5' d'un gène eucaryote, sur laquelle peuvent se fixer des facteurs transcriptionnels.
Caryotype
: Description du nombre et de la morphologie des chromosomes ; caractéristique d'une cellule, d'un individu ou d'une espèce.
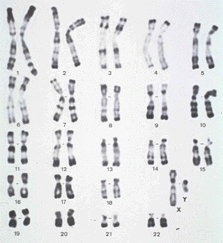
CARYOTYPE HUMAIN © - CNRS
cDNA
: Séquence de DNA complémentaire, synthétisée à partir de RNA grâce à une enzyme : la transcriptase inverse.
Centromère
: Région chromosomique identifiable sous forme d'une constriction ; point de départ des fibres du fuseau de division mitotique ou méiotique.
Chiasma
: Région cytologique identifiable dans les bivalents, correspondant au point d'échange de matériel génétique entre les chromatides d'une paire de chromosomes homologues (crossing-over).
Chimère
: Individu ou tissu constitué de deux populations cellulaires de génotypes différents, provenant de deux zygotes différents.
Chromatides
: Les deux éléments longitudinaux constituant chaque chromosome, visibles d'une part à partir du début de la prophase et surtout pendant la métaphase mitotiques, et d'autre part du diplotène à la métaphase de la deuxième division méiotique. Elles sont réunies par le centromère. Les chromatides soeurs sont les deux chromatides constituant un chromosome ; elles sont formées par réplication, contrairement aux chromatides non soeurs, issues de chromosomes homologues. Après le clivage des centromères pendant l'anaphase, on ne parle plus de chromatides, mais de chromosomes fils. Une cassure ou une aberration chromosomique chromatidienne n'affecte qu'une des deux chromatides soeurs. Elle se produit après la réplication du DNA, pendant la phase S. Une cassure survenue avant la phase S affecte les deux chromatides et est nommée anomalie isolocus (cassure isochromatidienne).
Chromatine
: Structure constitutive des chromosomes, associant DNA, protéines chromosomiques basiques (histones) et non histoniques et de petites quantités de RNA. Elle est visible dans les noyaux interphasiques.
Chromosome
: Support des gènes, il est constitué de chromatine, et visible pendant la division cellulaire sous forme de fins bâtonnets.
Clone
: Population de cellules ou d'organismes issus d'une même cellule ou d'un même ancêtre, qui lui sont identiques génétiquement et sont identiques entre eux.
Cluster de gènes
: Un groupe de deux ou plusieurs gènes situés sur un même chromosome, proches les uns des autres et dont les fonctions sont voisines (ex : gènes du système HLA ou des Immoglobulines).
CMH
: Complexe majeur d'histocompatibilité.
Codon
: Séquence de trois nucléotides de DNA ou de RNA codant pour un acide aminé ou pour le signal de terminaison d'une séqence d'acides aminés donnée.
Codon terminateur
: Un des signaux de ponctuation. Séquence de DNA signalant la fin de la transcription.
Coefficient de consanguinité
: Probabilité que, sur un locus donné du génome, les deux allèles homologues soient identiques par ascendance commune, c'est à dire qu'ils aient été hérités par la lignée paternelle et maternelle à partir d'un ancêtre commun aux deux parents.
Coefficient de sélection
: Mesure quantitative de la sélection.
Complémentation génétique
: Effet complémentaire d'une double mutation, se produisant sur des gènes différents. Les deux gènes mutés peuvent reconstituer un phénotype normal.
Concordance
: Survenue d'un caractère ou d'une maladie chez deux jumeaux monozygotes ou dizygotes.
Condensation chromosomique prématurée
: Induction d'une condensation chromosomique dans un noyau interphasique par fusion avec une cellule en mitose.
Conjugaison
: Transfert de DNA entre bactéries.
Consanguinité
: Est dit consanguin l'enfant issu de deux parents apparentés, c'est à dire ayant au moins un ancêtre en commun vérifiable ou vraisemblable.
Consensus (séquence)
: Courte séquence de DNA retrouvée dans différents gènes et différents êtres vivants.
Corpuscules de Barr
: Chromatine X.
Cosmide
: Vecteur plasmidique auquel, en plus des séquences nécessaires à sa multiplication normalement présentes, ont été rajoutés les sites cos du phage lambda. Il permet le clonage de grands fragments de DNA, jusqu'à 40 kb.
Croisement en retour (back-cross)
: Croisement entre un animal hétérozygote et l'un de ses parents. Le double croisement en retour (double test-cross) implique deux gènes, il s'agit du croisement entre un individu double hétérozygote et l'un de ses parents.
Crossing-over
: Echange réciproque d'information génétique entre deux chromosomes homologues, survenant au niveau des chiasmas pendant le stade diplotène de la méiose I et responsable d'une recombinaison génétique entre deux loci voisins (gènes liés).
Il existe non seulement des crossing-over méiotiques, mais également mitotiques se produisant pendant la mitose des cellules somatiques.
Cycle cellulaire
: Cycle de vie des cellules. On peut distinguer quatre phases successives dans le cycle de vie des cellules somatiques en division : la mitose, la phase G1, la phase S (réplication du DNA) et la phase G2. Les phases G1, S et G2 constituent l'interphase. Les cellules ne se divisant pas entrent en phase G0.

|
Déficience
: En génétique, anomalie de la structure chromosomique résultant de la perte d'un segment chromosomique.
Délétion
: Perte d'un segment chromosomique ou de DNA.
Dénaturation du DNA
: Transformation d'un double-brin de DNA en simple brin.
Dérive génétique (Drift)
: Variation aléatoire des fréquences alléliques dans une population. Ce phénomène, dû au hasard, joue un rôle important dans les populations de faibles effectifs. Le hasard intervenant dans la reproduction sexuée au cours de la méiose peut modifier la fréquence de la transmission d'un allèle d'une génération à une autre et donc provoquer, dans la population, une fluctuation de la fréquence de cet allèle d'une génération à une autre. Dans certaines conditions, un allèle peut totalement disparaitre de la population (élimination d'un allèle) ou être présent chez tous les individus (fixation d'un allèle).
Diacinèse
: Dernier stade de la prophase de la première division méiotique (prophase I).
Dicentrique
: Se dit d'un chromosome dont la structure est anormale car il comprend deux centromères.
Dictyotène
: Un des stades de la prophase de la première division méiotique, pendant lequel l'ovogenèse des mammifères s'interrompt. Dans l'espèce humaine, les ovocytes atteignent le stade dictyotène environ 4 semaines avant la naissance et restent bloqués à ce stade jusqu'à l'ovulation. La méiose ne se poursuit que lors de l'ovulation.
Diploïde
: Cellule ou organisme possédant deux jeux de chromosomes homologues, l'un d'origine paternelle et l'autre d'origine maternelle.
Diplotène
: Un des stades de la prophase de la première division méiotique (prophase I).
Discordance
: Non-identité d'un caractère (ex : une maladie entre deux jumeaux).
Disomie uniparentale (DUP)
: Situation dans laquelle les deux chromosomes constituant une paire chromosomique proviennent d'un seul des parents.
Dispermie
: Fécondation d'un ovule par deux spermatozoïdes.
Dizygote
: Se dit de jumeaux bi-ovulaires, provenant de deux oeufs différents, contrairement aux jumeaux monozygotes (uni-ovulaires).
DNA (acide désoxyribonucléique)
: La molécule de DNA constitue le matériel génétique. Elle est composée de deux chaînes polynucléotidiques. L'information génétique y est disposée sous forme d'une séquence linéaire, constituée d'un enchaînement de trinucléotides (triplet ou codon).
DNA anti-sens
: Un brin de DNA complémentaire d'un RNA messager et servant de matrice (template) à la transcription.
DNA recombiné
: Une molécule de DNA constituée de parties de différentes origines.
DNA satellite (sDNA)
: Le sDNA se distingue de la majeure partie du DNA par son assemblage de bases. Après centrifugation en gradient continu de chlorure de césium, le sDNA apparaît sous forme de bandes individualisables (satellites), de densité inférieure à celle caractérisant la majeure partie du DNA. Cette différence de densité est due à la différence de poids moléculaire, elle-même due à la différence de l'assemblage des bases les constituant.
Ne pas confondre le sDNA et la région satellite des chromosomes acrocentriques.
Dominant
: Se dit d'un gène ou d'un allèle qui s'exprime à l'état hétérozygote. La notion de dominance et de récessivité ne se rapporte qu'à l'effet phénotypique d'un gène donné. Deux allèles s'exprimant conjointement à l'état hétérozygote sont dits codominants.
Dosage génique
: Nombre de gènes fonctionnels (exprimés) dans un génôme.
DUP
: Disomie uniparentale (voir Disomie).
Duplication
: Modification de la structure d'un chromosome, responsable du dédoublement d'un segment chromosomique ou de quelques paires nucléotidiques. La duplication d'un gène (duplication génique) a joué un rôle essentiel dans l'évolution des eucaryotes.

|
Empreinte parentale (ou génomique)
: Non-identité de l'expression d'un allèle ou d'un segment chromosomique selon son origine parentale.
Endonucléase
: Classe hétérogène d'enzymes pouvant cliver la chaîne polypeptidique et intervenant donc dans le catabolisme des acides nucléiques.
Endoréplication
: Réplication chromosomique pendant l'interphase non suivie d'une mitose normale. Les chromosomes endorépliqués peuvent être identifiés pendant la métaphase, car ils sont constitués de quatre chromatides situées les unes contre les autres, unies par deux centromères très proches.
Enhancer
: Séquence régulatrice (stimulatrice) de DNA, renfermant des sites de fixation de facteurs transcriptionnels. Les séquences enhancer sont situées à une distance variable du promoteur. Elles augmentent considérablement le taux de transcription, le multipliant environ par dix.
Enzyme
: Protéine capable, par des propriétés catalytiques, d'activer une réaction chimique donnée. Les enzymes sont constituées d'une partie protéique (apo-enzyme), leur conférant leur spécificité, et d'une partie non protéique (coenzyme), participant à la réaction. Les enzymes se lient aux substrats. Après la réaction chimique, le substrat est modifié ou se lie à d'autres substances.
Enzyme de restriction (endonucléase de restriction)
: Endonucléase clivant spécifiquement le DNA au niveau d'une séquence nucléotidique spécifique.
Episome
: Plasmide pouvant exister isolément de manière autonome ou être inséré dans le génôme de l'hôte bactérien.
Epissage (splicing)
: Maturation des transcrits primaires (RNA pré-messagers) par excision des introns et rabouttage (épissage) des exons situés de part et d'autre de chaque intron excisé.
Epistasis
: Interaction entre gènes situés sur un même locus (allélique) ou sur différents loci (non allélique), résultant en une modification de l'expression phénotypique.
Espèces
: Populations naturelles constituées d'individus pouvant se reproduire entre eux et formant un pool génique commun.
Eucaryote
: Plante ou animal dont les cellules possèdent un noyau renfermant des chromosomes et se divisent par mitose ou méiose (contrairement aux procaryotes : virus, bactéries et algues bleu-vert, qui ne présentent pas les caractéristiques de l'organisation génétique des eucaryotes).
Euchromatine
: Chromosomes ou segments chromosomiques non individualisables dans les cellules interphasique. En effet, ils ne sont identifiables par des colorations spécifiques qu'au cours de la mitose (voir Hétérochromatine). L'euchromatine correspond à la partie génétiquement active, non condensée de la chromatine des noyaux cellulaires interphasiques.
Euploïde
: Se dit d'une cellule, d'un tissu ou d'un individu présentant le nombre normal de chromosomes, ce nombre caractérisant chaque espèce.
Exon
: Un segment de DNA codant.
Exonucléase
: Classe d'enzyme catalysant l'hydrolyse des acides nucléiques situés aux extrémités (voir Endonucléase).
Expression
: Désigne l'effet d'un allèle situé sur un locus donné. En présence de cet effet, on dit que l'allèle s'exprime.
Expressivité
: Désigne le type ou l'intensité de l'expression phénotypique d'un gène ou d'un génotype. On parle de défaut de pénétrance en présence d'un défaut de l'expression d'un allèle.

|
Fourche de réplication
: Région déroulée de la double hélice de DNA où se produit la réplication.
Fragments d'Okazaki
: Courtes séquences nucléotidiques synthétisées lors de la réplication discontinue d'un des brins de DNA et finalement liées les unes aux autres.
Fréquence allélique (ou génique)
: Fréquence d'un allèle donné par rapport à l'ensemble des allèles existant dans la population, pour un gène déterminé.
Fréquence de recombinaison
: Mesure de la distance génétique entre deux ou plusieurs loci.

|
Gamète
: Cellule germinale : spermatozoïde (dans le sexe masculin) ou ovule (dans le sexe féminin). Chez les mammifères, le sexe masculin est hétérogamétique (XY) et le sexe féminin est homogamétique (XX). Par contre chez les oiseaux, le sexe féminin est hétérogamétique (ZW) et le sexe masculin est homogamétique (ZZ).
Gen-flow
: Propagation des allèles d'une population à une autre.
Gène
: Facteur héréditaire. Il constitue l'unité de fonction du génome. Il s'agit d'un segment de DNA codant pour une chaïne polypeptidique (voir Cistron).
Génétique
: (dérivé du grec genesis : genèse, origine) Science de l'hérédité, qui étudie la transmission des caractères et les différences d'origine génétique distinguant les organismes.
Génome
: Ensemble du matériel génétique d'une cellule ou d'un individu. Désigne généralement le lot haploïde de chromosomes.
Génotype
: Ensemble de l'information génétique d'un individu ou d'une cellule.
Germinal
: Se dit des gamètes et de leurs précurseurs (cellules germinales), par opposition aux cellules somatiques.
Globule polaire
: Cellule formée pendant l'ovogenèse, qui ne se transforme pas en ovule mais dégénère.

|
Haploïde
: Cellules ou individus possédant un seul jeu chromosomique ; les gamètes sont des cellules haploïdes.
Haplotype
: Combinaison d'allèles de deux ou plusieurs loci différents mais proches sur un même chromosome (liés). (ex : les allèles du système HLA).
Hémizygote
: Se dit des gènes et des loci présents en un seul exemplaire dans le génotype. Ex : les gènes et les loci situés sur le chromosome X dans le sexe masculin (XY).
Hétérocaryon
: Cellule binucléaire ou multinucléaire, possédant au moins deux noyaux. Elle est obtenue par fusion de cellules de même espèce ou d'espèces différentes et dont le génotype diffère.
Hétérochromatine
: Chromosomes ou segments chromosomiques visibles pendant la fin de la télophase, l'interphase et le début de la prophase sous forme d'une structure compacte et colorée, contrairement à l'euchromatine visible uniquement dans les cellules en division (voir Euchromatine). L'hétérochromatine correspond à des chromosomes ou des segments chromosomiques génétiquement inactifs ou peu actifs. On peut différencier l'hétérochromatine constitutive et facultative.
Hétérodisomie
: Situation dans laquelle une paire de chromosomes est constituée de deux chromosomes différents, provenant d'un seul des deux parents (voir Isodisomie).
Hétéroduplex
: Molécule de DNA constituée de deux brins d'origine chromosomique ou de nature différente et comprenant une région non complémentaire, c'est-à-dire présentant un mésappariement.
Hétérogamétique
: Situation dans laquelle deux différents types de gamètes sont fomés, p.ex. X et Y chez les mammifères (voir Gamètes).
Hétérogénéité génétique
: Deux ou plusieurs génotypes s'expriment par un même phénotype.
Hétéroploïdie
: Anomalie du nombre de chromosomes, présents dans des cellules ou des tissus isolés.
Hétérosis
: Avantage des génotypes hétérozygotes par rapport aux génotypes homozygotes, chez des plantes et des animaux.
Hétérozygotie
: Présence de deux allèles différents sur les deux loci homologues d'une même paire chromosomique (voir Homozygote).
Histocompatibilité
: Compatibilité tissulaire, dérterminée par le complexe majeur d'histocompatibilité (CMH).
Histones
: Protéines chromosomiques basiques présentes dans les chromosomes des eucaryotes.
HLA (Human Leukocytes Antigen)
: Principal complexe d'histocompatibilité chez l'homme.
Homeobox
: Séquence de DNA très conservée, retrouvée dans les gènes homéotiques (essentielle au développement embryonnaire).
Homologue
: En génétique, désigne les deux chromosomes d'une même paire chromosomique et les loci qui y sont situés, un des chromosomes étant d'origine maternelle et l'autre d'origine paternelle.
Homozygotie
: Présence du même allèle sur les deux loci homologues d'une même paire chromosomique.
Hybridation
: Croisement entre des animaux ou des plantes de génotypes différents, appartenant ou non à une même espèce. En génétique, ce terme est utilisé pour désigner deux situations différentes :
(1) en biologie moléculaire, appariement de deux séquences nucléotidiques complémentaires, soit deux brins de DNA (hybridation DNA/DNA), soit un brin de DNA et un brin de RNA (hybridation DNA/RNA).
(2) en cytogénétique, fusion de deux cellules ayant un génotype différent, issues de la même espèce ou d'espèce différentes (hybridation cellulaire).

|
Insertion
: Intégration dans un chromosome de matériel génétique d'origine non homologue (voir Translocation).
Intron
: Séquence de DNA non codante, située dans un gène (voir Exon).
Inversion
: Anomalie de la structure chromosomique résultant de deux cassures se produisant dans des régions différentes d"un même chromosome, avec changement d'orientation du segment intercalaire puis recollement de ce segment. On peut distinguer deux types d'inversions : péricentriques et paracentriques.
Dans l'inversion péricentrique, le centromère est situé dans le segment inversé. Il en résulte une modification de la position du centromère.
Dans l'inversion paracentrique, le centromère n'est pas situé dans le segment inversé. Ce type d'inversion ne modifie pas la position du centromère ; la forme du chromosome y est donc inchangée.
Les inversions chromosomiques jouent un rôle important dans l'évolution caryologique.
Isochromosome
: Chromosome constitué de deux bras identiques, longs ou courts, p. ex. isochromosome X. Il résulte de la perte d'un bras chromosomique et de la duplication du bras restant. Un isochromosome peut posséder un ou deux centromères.
Isodisomie
: Situation dans laquelle une paire de chromosomes est constituée de deux chromosomes identiques, provenant d'un seul des deux parents (voir Hétérodisomie).
Isolat génétique
: Population d'effectif faible n'ayant pas ou que peu d'échanges génériques avec l'extérieur (reproduction en milieu fermé) en raison d'un isolement géographique ou social (pas de panmixie).
Isozymes ou isoenzymes
: Enzymes de structure différentes les unes les autres, mais de jonctions voisines, dans un même organisme. Les esozymes sont l'expession biochimique d'un polymorphisme génétique.

|
Leptotène
: Un des stades de la méiose (voir Médiose).
Liaison génétique
: Situation dans laquelle deux loci proches sur un même chromosome, peuvent coségréger et donc être hérités ensemble (gènes liés). Le degré de liaison est mesurable, il depend de la distance génétique.
LINE (Long Interspersed Nuclear Element)
: Longues séquences répétées de DNA, dispersées dans le génome.
Linker
: Séquence de DNA bicaténaire synthétique, contenant un site de clivage pour une enzyme de restriction et pouvant lier deux fragments de DNA.
Locus
: Position d'un gène sur un chromosome ou dans un groupe de liaison.
Lymphocyte
: Cellule du système immunitaire, nettement différenciable en lymphocyte B issu de la moelle osseuse et lymphocyte T thymodépendant.
Lysosome
: Particule cytoplasmique contenant des enzymes hydrolasiques.

|
Marquage en bandes
: Coloration spécifique des chromosomes, provoquant l'apparition de bandes claires et sombres, perpendiculaires à l'axe chromosomique, et correspondant à différents segments chromosomiques. Le marquage en bandes est spécifique de chaque segment chromosomique d'un même chromosome. Les différents segments se distinguent en fonction du nombre, de la longueur, de la position relative et de l'intensité des bandes transversales sombres et pâles. Le marquage en bandes peut donc servir à l'identifiction des segments chromosomiques. Initialement, la notion de bandes fut introduite par Painter en 1939, les chromosomes polytènes de certains diptères (moustiques, mouches) étant formés d'une succession de bandes linéaires claires et sombres. Chaque bande est définie par rapport aux bandes voisines. Les bandes claires sont nommées interbandes.
Marqueur génétique
: Trait génétique héréditaire, utile au marquage d'un locus voisin.
Mb
: Mégabase. Correspondant à 1 million de paires de bases.
Méiose
: Division cellulaire et donc nucléaire, particulière, se produisant dans les cellules germinales et résultant en la réduction du jeu diploïde de chromosomes en un jeu haploïde. La prophase de la première division méiotique est constituée de plusieurs stades : leptotène, zygotène, pachytène, diplotène et diacinèse, et est particulièrement importante.
Métacentrique
: Se dit d'un chromosome dont les deux bras sont de même longueur, en raison de la position médiane du centromère.
Métaphase
: Deuxième phase de la mitose, pendant laquelle les chromosomes sont nettement visibles sous forme de fins bâtonnets.
Mitose
: Division cellulaire et donc nucléaire survenant dans les cellules somatiques. Elle comprend plusieurs phases : la prophase, la métaphase, l'anaphase et la télophase.
Mixoploïdie
: Tissus ou individus dont les cellules ont des caryotypes différents (mosaïque chromosomique).
Monosomie
: Perte d'un ou plusieurs chromosomes (monosomie complète) ou de segments chromosomiques (monosomie partielle), dans un jeu chromosomique diploïde. Dans l'espèce humaine, la seule monosomie complète non létale est la monosomie X, qui se traduit par le syndrome de Turner.
Monozygote
: Se dit de jumeaux provenant de la division anormale d'un oeuf unique (voir Dizygote).
Morgan
: Unité de distance génétique, constituée de 100 centimorgan (cM). La distance entre deux loci, exprimée en morgans, correspond à la fréquence de recombinaison entre ces deux loci. Nommé d'après Thomas H. Morgan (1866-1945), fondateur des études génétiques sur la drosophile.
Mosaïque
: Tissus ou individus constitués de cellules génétiquement différentes, bien qu'en principe issues d'un même zygote.
Mutagène
: Se dit d'une substance chimique pouvant induire une mutation.
Mutation
: Modification du matériel génétique, transmissible aux générations suivantes, c'est à dire modification de la séquence de DNA. On peut distinguer la mutation ponctuelle, la délétion ou l'insertion de paires de bases dans un gène, et la mutation chromosomique résultant de la modification de la structure chromosomique.

|
Non-disjonction
: Anomalie de la ségrégation d'une paire de chromosomes homologues lors de la méiose. Si l'anomalie de ségrégation survient lors de la mitose, il s'agit d'une non-disjonction mitotique.
Nucléoside
: Liaison d'une base purique ou pyrimidique et d'un sucre (ribose ou désoxyribose, voir Nucléotide).
Nucléosome
: Structure de base de la chromatine.
Nucléotide
: Elément constitutif des acides nucléiques. En effet, les acides nucléiques sont des polymères de nucléotides. Un nucléotide est un ester phosphorique constitué d'une base purique ou pyrimidique, d'un pentose (ribose ou désoxyribose) et d'un radical phosphate.

|
Oncogène
: Gène dont la présence induit une transformation maligne.
Opérateur
: Une des séquence régulatrices présentes dans certains opérons. La fixation d'un répresseur sur l'opérateur empêche toute transcription du gène.
Opéron
: Unité d'expression constituée de gènes de structure, regroupés sur le DNA (liés) codant pour des protéines impliquées dans une même séquence métabolique et pouvant être corégulés, et de séquences régulatrices.

|
Paire de bases
: Se dit de deux bases puriques ou pyrimidiques situées face à face dans la molécule de DNA.
Palindrome
: Séquences nucléotidiques répétitives inversées, pouvant former des structures en forme d'épingles à cheveux, par repliement d'une chaîne de DNA linéaire monocaténaire.
Panmixie
: Situation dans laquelle les fréquences des différents types de croisements possibles dans une population sont égales, le choix des conjoints s'effectuant au hasard, contrairement au croisement préférentiel.
PCR
: Polymerase Chain Reaction ou amplification génique in vitro par la DNA-polymérase. Technique permettant la multiplication (ou amplification) in vitro d'une séquence de DNA donnée par la répétition de cycles de réplication du DNA. Chaque cycle est constitué de trois étapes, nécessitant chacune une température différente : (1) dénaturation, (2) hybridation avec les oligonucléotides de synthèse spécifiques de la séquence cible et qui servent d'amorce (ou primer), et (3) élongation de la chaîne.
Pénétrance
: Probabilité ou fréquence de l'expression d'un gène (voir expressivité).
Peptide
: Substance formée de deux ou plusieurs acides aminés unis par une liaison peptidique.
Phénotype
: Manifestation apparente du génotype d'une cellule ou d'un individu ou de la répartition d'une fréquence donnée dans une population. Elle résulte de l'interaction de facteurs génétiques et du milieu extérieur.
Plasmide
: Fragment de DNA extra-chromosomique le plus souvent circulaire, indépendant ou inséré dans le chromosome hôte.
Pléiotropie
: Situation dans laquelle un gène s'exprime par de multiples phénotypes, en apparence indépendants les uns des autres.
Polygénique
: Se dit d'un caractère ou d'un mode de transmission de l'hérédité sous la dépendance de plusieurs gènes. Les effets individuels de ces gènes ne sont pas décelables.
Polymérases
: Enzymes catalysant la réaction liant les nucléotides dans le RNA ou le DNA (réplication du DNA ou transcription).
Polymorphisme génétique
: Présence de plusieurs allèles sur un ou de nombreux loci, s'exprimant par différents phénotypes. Par convention, on parle de polymorphisme lorsque chaque phénotype est présent chez au moins 1% de la population.
Polyploïde
: Cellules, tissus ou individus caractérisés par la présence de plus de deux jeux chromosomiques (diploïde) : trois (triploïde), quatre (tétraploïde), cinq (pentaploïde) ou plus. Chez l'homme, la triploïdie et la tétraploïdie constituent le plus souvent un facteur de létalité et provoquent en principe un avortement spontané.
Polytène
: Se dit d'un type particulier de chromosomes formés par endoréplication successives de chromatides. Ceci résulte en des chromosomes géants.
Population
: Ensemble d'individus appartenant à une même espèce, défini comme un groupe de reproduction tel que la plupart des individus choisissent leur conjoint dans le groupe, et formant un pool génétique commun.
Procaryote
: Organisme unicellulaire dépourvu de noyau et d'organite cellulaire.
Promoteur
: Région du DNA intervenant dans la régulation de la transcription, car comportant le site de fixation de la RNA-polymérase.
Prophage
: Génome viral intégré dans le génome bactérien.
Prototrophe
: Bactérie se multipliant indépendamment des substances présentes dans le milieu de culture.

|
|
Quadrivalent
: Structure, généralement en forme de croix, formée de chromosomes appariés homologues ou non homologues, visible lors d'une translocation survenant pendant une division mitotique.
|
Race
: Les races sont des populations de même espèce qui se différencient les unes des autres par la fréqence de quelques-uns de leurs gènes. En conséquence, la notion de race est variable et définie en fonction de la phylogenèse.

|
|
|